Cuando inicia sesión en LinkedIn, generalmente se le presentan sugerencias para conectarse con personas que conoce, fue porque fue a la misma universidad que ellos o que trabajó en la misma empresa o industria.
Sin embargo, las sugerencias a veces pueden sorprendernos, como cuando el algoritmo recomienda un primo o un amigo de la familia a pesar de que lo hacen en un campo completamente diferente. Dada la falta general de superposición profesional, es posible que se pregunte cómo Linkedn podría saber sobre estas relaciones de la vida real.
Algoritmos de inteligencia artificial (AI) que ejecutan estas recomendaciones utilizan el tipo de tecnología conocida como una red neuronal gráfica basada en gráficos: estructuras matemáticas hechas de nodos y conexiones (también conocidas como “bordes”) que los conectan. Para una red social como LinkedIn, el gráfico se puede generar donde los nodos representan a cada usuario, mientras que los enlaces están conectados entre ellos.
Estos algoritmos recopilan información de la vecindad inmediata de cada nodo: nuestros enlaces directos en LinkedIn. Luego los agregan e integran en el nodo original.
Después de este proceso, cada perfil refleja sus propios datos y la red inmediata. Este proceso se puede informar varias veces: en otra iteración, cuando recopilemos información de nuestros vecinos, ya tendrán información recopilada de sus propios vecinos y, por lo tanto, tendremos información de otro entorno.
Un ejemplo de una red social. El nudo morado representa el perfil de LinkedIn. Las conexiones inmediatas (vecinos de primer grado) se extienden al exterior, con vecinos del segundo y tercer grado más. М. Hernaez / Bienender. Relaciones web
En estas redes, no solo nuestra información personal es importante, sino también con quién hemos conectado y con quién están conectados con nosotros. En la versión completa del algoritmo de LinkedIn, como se usa en la práctica, no solo hay nodos que representan a las personas, sino también a otros tipos de nodos, como empresas o publicaciones.
Esto significa que el algoritmo puede obtener información de nuestras conexiones personales y marcamos el contenido como favoritos o comunicados.
Si, por ejemplo, alguien tiene a su hermana como conexión y “le gustaron” las publicaciones que le encantan a su yerno, el algoritmo no solo puede compartir los intereses, sino que de alguna manera puede de alguna manera.
Algoritmos de redes sociales en biomedicina
Desarrollar un medicamento a partir de cero es extremadamente costoso y prolongado. El proceso de descubrimiento a menudo recuerda el embudo. En la parte superior, todos los candidatos potenciales ingresan y, después de estrecharse en varias etapas de investigación, solo quedó uno para ingresar ensayos clínicos. Este medicamento (con suerte) irá a estar disponible para uso clínico entre la población general.
Aunque es necesario, la complejidad de esta verificación significa que el cambio de drogas se ha convertido en común en común durante décadas. El objetivo de este proceso no es diseñar nuevos medicamentos, sino encontrar un nuevo uso para los existentes.
Trate la enfermedad, nos centramos principalmente en dirigir las proteínas responsables de eso. Existen bases de datos públicas y bien documentadas que contienen información sobre qué proteínas de cualquier objetivo de drogas y esta base de datos han aumentado significativamente en los últimos años.
Una de las bases de datos más utilizadas, DRGBank, excedió 841 medicamentos aprobados cuando se publicó por primera vez en 2006. Años, en 2.751 en la última actualización 2024. Años. Esta creciente disponibilidad de datos proporciona el uso de modelos más complejos.
Con esta cantidad de datos, podemos crear una red de gráficos donde los medicamentos y las proteínas son nodos, y los lazos interactúan entre ellos, como se registran en bases de datos. Una vez que tenemos una red, podemos aplicar algoritmos similares a los utilizados en las redes sociales: para cada fármaco, agregamos información bioquímica sobre proteínas para comunicarse a través de enlaces conocidos.
Usando esta información, el modelo puede decirnos la probabilidad de interacción en la proteína de drogas que no hemos tenido en la base de datos antes, porque los algoritmos pueden analizar efectivamente grandes cantidades de información. Estas interacciones pueden confirmarse en condiciones de laboratorio, ahorrando tiempo y dinero de un proceso de detección de larga duración.
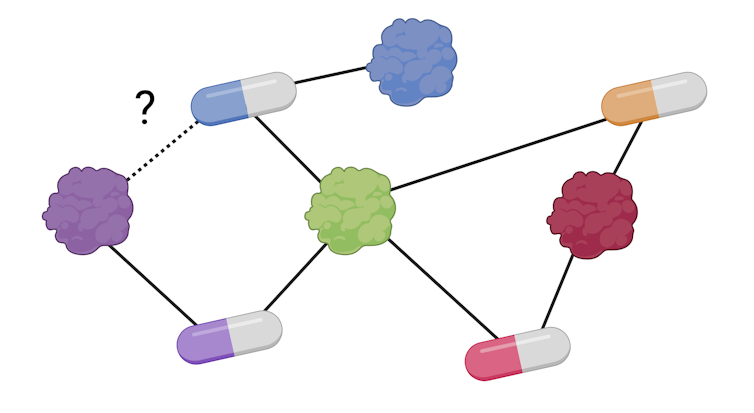
La interacción de red de la medicación proteica. Los enlaces en negro son interacciones conocidas. La marca de designación en la línea intermitente indica la interacción de la cual queremos confirmar. М. Hernaez / Bioreder nuestra investigación
En Biología informática y Laboratorio de Traducción de la Universidad de Navarra, seguimos esta idea para desarrollar Gennius, un modelo que tiene como objetivo construir una red entre medicamentos y proteínas. Su aplicación ya ha mejorado los modelos existentes, especialmente en términos de tiempo de trabajo: en solo un minuto podemos evaluar unas 23,000 interacciones.
Si bien el modelo tiene buenas posibilidades predictivas, todavía hay margen de mejora. Por ejemplo, aparecen desafíos al evaluar posibles interacciones con moléculas que no son parte de la red o para las cuales tenemos datos originales pequeños. Aunque técnicamente es posible generar la salida, el modelo a menudo ofrece resultados de confianza más bajos en estos casos.
Al superar estos obstáculos y más investigaciones, estos modelos podrían desarrollarse en sistemas futuros que proporcionan recomendaciones personalizadas para cada paciente.
Descubre más desde USA Today
Suscríbete y recibe las últimas entradas en tu correo electrónico.









